MLZ ist eine Kooperation aus:
 > Technische Universität München
> Technische Universität München > Helmholtz-Zentrum Hereon
> Helmholtz-Zentrum Hereon
 > Forschungszentrum Jülich
> Forschungszentrum Jülich
MLZ ist Mitglied in:
 > LENS
> LENS > ERF-AISBL
> ERF-AISBL
MLZ in den sozialen Medien:

MLZ
Lichtenbergstr.1
85748 Garching
Neutronen können Proteinen bei der Arbeit zusehen
Proteine sind biologische Makromoleküle und finden sich in allen Zellen. Dort sorgen sie für Festigkeit oder ermöglichen als molekulare Maschinen Stoffwechselprozesse, etwa den Transport oder die Nutzbarmachung von Energieträgern. Die Funktion mancher Proteine beruht auf der passgenauen Erkennung eines Substrates und der Umformung der Proteinstruktur, um das Produkt herstellen zu können. Eine Reihe von Neutronenstreumethoden bietet einzigartige Möglichkeiten, die Struktur und Bewegung der Proteinbereiche während ihrer Arbeit zu erkennen.
Proteine sind langkettige Makromoleküle. Sie bestehen aus Ketten von Aminosäuren einer spezifischen Reihenfolge und charakteristischer dreidimensionaler Anordnung. Beides gemeinsam bestimmt ihre spezifischen chemischen oder physikalischen Eigenschaften. Eine Besonderheit vieler Proteine ist das „aktive Zentrum“, in dem ein katalytischer Prozess stattfindet. Dies ist ein Bereich, an dem ein genau passendes kleines Molekül andocken kann, das „Substrat“. Dies kann einen strukturellen oder chemischen Prozess auslösen. Die strukturellen Prozesse reichen von der Umstellung einzelner Atome bis zur Neuanordnung kompletter Untereinheiten der Proteine, so genannter „Domänen“ Sie dienen dazu Substrate einzuschließen, Produkte freizusetzen oder Domänen umzugestalten.
Die Neutronenstreuung bietet einzigartige Möglichkeiten, um solches Verhalten von Proteinen in ihrer annähernd natürlichen wässrigen Umgebung zu untersuchen: So lässt sich mittels Kleinwinkelstreuung oder kurz KWS die Anordnung und Form von Domänen erkennen. Neutronenspinecho-Spektroskopie (NSE) ermöglicht die Beobachtung der Bewegung auf den relevanten Längen- und Zeitskalen (Nanometer und Nanosekunden), mit denen sich komplette Domänen dynamisch umorientieren. Neutronenkristallographie an Proteinkristallen mit dem Instrument Biodiff erlaubt es, Wasserstoffatome bis zu einer Auflösung von 0.2 Nanometer zu lokalisieren, im Gegensatz zur Röntgenkristallographie, die die Elektronendichteverteilung nachweist und Wasserstoffatome nicht sieht. Die Rückstreutechnik schließlich detektiert Atombewegungen in Größenordnungen von zwischenatomaren Distanzen und Pikosekunden.
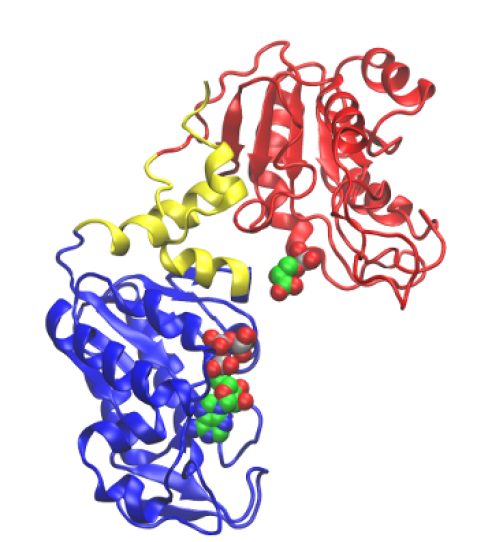
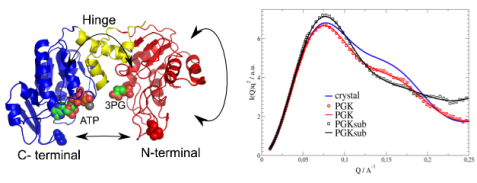
Am Beispiel der Phosphoglyceratkinase1 (PGK [1], Abb. 1) lässt sich erkennen, wie verschiedene Neutronenstreumethoden ineinandergreifen. Die PGK katalysiert einen Schritt in der Glykolyse, einem Stoffwechselpfads, der in Zellen Energie aus Zucker erzeugt. Die PGK besteht aus zwei weit geöffneten Domänen, die über ein Gelenk in der Nähe des aktiven Zentrums miteinander verbunden sind. In beiden Domänen findet sich jeweils eine besonders wichtige Aminosäure des aktiven Zentrums, welche eine Schlüsselfunktion beim Transfer einer Phosphatgruppe ausüben.
Strukturanalysen mittels KWS (Abb. 2) wiesen nach, dass die PGK in Lösung zwar eine kompaktere Form besitzt als das kristallisierte Protein, aber die Distanz zwischen den beiden Schlüsselaminosäuren dennoch zu groß ist, um die Übertragung der Phosphatgruppe zu ermöglichen. Experimente mit NSE zeigten thermisch getriebene, großräumige Molekularbewegungen der PGK-Domänen in einem Zeitbereich von 50 Nanosekunden. Könnten diese intrinsischen Fluktuationen des Proteins stark genug sein, um das Protein in eine reaktive Form zu bringen, bei der die zwei Schlüsselaminosäuren nah genug zusammenkommen, um eine Reaktion des gebundenen Substrats auszulösen?
Ein Vergleich der gemessenen Dynamik mit den langsamsten Bewegungsmustern einer Modenanalyse bestätigte diese Vermutung. Eine Normalmodenanalyse berechnet alle möglichen Bewegungen eines Proteins, basierend auf Informationen über die atomare Struktur. Die langsamsten berechneten Bewegungen, welche die größten Auslenkungen besitzen, zeigten eine nahezu perfekte Übereinstimmung mit der gemessenen Dynamik. Da diese Bewegung die Schlüsselaminosäuren nah genug zueinander und zum aktiven Zentrum bringt, um die aktive Form des Proteins zu bilden, ermöglichen die beobachteten Bewegungen tatsächlich die Funktionsfähigkeit des Proteins.
Referenzen
[1] Inoue et al Biophysical Journal, 2010, 99, 2309 – 2317
Originalpublikation:
N. Smolin, R. Biehl, G.R. Kneller, D. Richter & J.C. Smith.; Functional Domain Motions in Proteins on the ~1-100ns Timescale: Comparison of Neutron Spin Echo Spectroscopy of Phosphoglycerate Kinase with Molecular Dynamics Simulation; Biophys J. 2012 Mar 7; 102(5):1108-17. DOI: 10.1016/j.bpj.2012.01.002. Epub 2012 Mar 6.
MLZ ist eine Kooperation aus:
 > Technische Universität München
> Technische Universität München > Helmholtz-Zentrum Hereon
> Helmholtz-Zentrum Hereon
 > Forschungszentrum Jülich
> Forschungszentrum Jülich
MLZ ist Mitglied in:
 > LENS
> LENS > ERF-AISBL
> ERF-AISBL
MLZ in den sozialen Medien: