MLZ ist eine Kooperation aus:
 > Technische Universität München
> Technische Universität München > Helmholtz-Zentrum Hereon
> Helmholtz-Zentrum Hereon
 > Forschungszentrum Jülich
> Forschungszentrum Jülich
MLZ ist Mitglied in:
 > LENS
> LENS > ERF-AISBL
> ERF-AISBL
MLZ in den sozialen Medien:

MLZ
Lichtenbergstr.1
85748 Garching
16.10.2020
Der Zauber liegt im Detail: Leuchtprotein exakter entschlüsselt

Wissenschaftler des Heinz Maier-Leibnitz Zentrums haben den Kristall des Leuchtproteins am Instrument BioDiff untersucht.
Das Grün fluoreszierende Protein GFP ist von immenser Bedeutung, als leuchtender Kundschafter in der Zelle. Am Heinz Maier-Leibnitz Zentrum (MLZ) untersuchten japanische Forscherinnen und Forscher die Position der Wasserstoffatome in den farbgebenden Bestandteilen des Eiweißes, was die Entwicklung neuer markierender Proteine ermöglicht.
Fluoreszierende Proteine sind ein wichtiges Werkzeug in der Biologie. Forscher können sie mit anderen Proteinen koppeln. Das grün fluoreszierende Protein (GFP) ist dabei das bekannteste Fluoreszenzmolekül. Bestrahlt man es mit blauem Licht, leuchtet es grün. So fungiert GFP als Marker für das Molekül, mit dem es gekoppelt ist. Das macht es Forschenden möglich, die räumliche und zeitliche Verteilung von Proteinen auch im lebenden Organismus direkt zu beobachten: Sie verfolgen einfach das grüne Licht unter dem Mikroskop.
Japanischen Wissenschaftlerinnen und Wissenschaftlern um Dr. Motoyasu Adachi von den National Institutes for Quantum and Radiological Science and Technology in Ibaraki gelang es am Instrument BioDiff des MLZ, die Struktur des Leuchtproteins an entscheidenden Stellen genauer zu entschlüsseln. Sie untersuchten die An- und Abwesenheit von Wasserstoffatomen (Protonen) in einer Mutante des GFP, namens EGFPq. Diese Version weist gezielte Mutationen an mehreren Stellen auf, die die Fluoreszenz und Stabilität des Proteins verstärken. Die neuen Erkenntnisse der Forscherinnen und Forscher können bei dem Design zukünftiger Marker-Proteine Anwendung finden.
Neutronen machen die Wasserstoffbrücken-Netzwerke in farbgebenden Teilen sichtbar
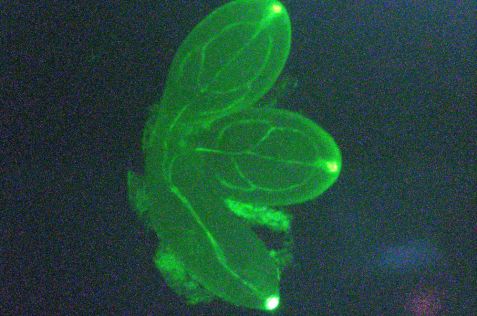
Das grün fluoreszierende Protein dient als Marker in der Gentechnik, wie hier in den Keimblättern der Acker-Schmalwand Arabidopsis thaliana. Es zeigt an, wo ein bestimmtes Eiweiß in der Pflanze hergestellt wird.
Das Instrument BioDiff am MLZ ist darauf ausgelegt, die Struktur von biologischen Makromolekülen wie Proteinen zu bestimmen. Wissenschaftlerinnen und Wissenschaftler können hier vor allem die Position der besonders leichten Wasserstoffatome untersuchen, was mit anderen gängigen Methoden wie der Röntgenstrukturanalyse nur in Ausnahmefällen möglich ist.
„Ein Vorteil des monochromatischen Einkristall-Diffraktometers BioDiff ist die hohe Auflösung, die wir erreichen können“, erklärt Dr. Andreas Ostermann von der Technischen Universität München. Gemeinsam mit seinem Kollegen Dr. Tobias Schrader vom Forschungszentrum Jülich und ebenfalls Instrumentwissenschaftler am BioDiff betreute er die japanischen Wissenschaftler. „Mit dem jetzt gemessenen Datensatz in einer Auflösung von 1,45 Ångström, dem zehnmillionsten Teil eines Millimeters, sind wir unter den fünf besten Datensätzen in der Neutronen-Diffraktion für Proteine mit mehr als 100 Aminosäuren“ Die Forscher konnten mithilfe der Neutronenkristallographie am BioDiff die charakteristischen Protonierungszustände der farbgebenden Bestandteile des GFP und umliegender Schlüsselstrukturen analysieren.
Noch exakter als Messungen mit Röntgenstrahlen
Die Wissenschaftlerinnen und Wissenschaftler bestimmten die Position der Protonen im aktiven Zentrum des GFP mittels der Neutronen-Diffraktion exakter als jemals zuvor. Dabei stellten sie minimale Abweichungen in den Bindungslängen zwischen den Wasserstoffatomen und dem Sauerstoffatom eines zentralen Wassermoleküls im aktiven Zentrum fest. „Das Wasser ist also ein bisschen verzerrt“, erklärt Ostermann. „Unsere japanischen Kollegen gehen davon aus, dass diese Verzerrung möglicherweise durch ein ungewöhnliches elektrostatisches Potential an dieser Stelle ausgelöst wird.“
Die spannendste Abweichung liegt im Baustein namens „His148“: Diese Struktur sollte flach in der Ebene liegen – tut sie aber nicht. Diese Abweichung konnte man bisher mit Röntgenstrahlung nicht nachweisen.
Die Ergebnisse könnten dazu beitragen weitere maßgeschneiderte Markerproteine zu designen. Oft geschieht dies auf Grundlage quantenchemischer Berechnungen. Hierfür verwenden Proteindesigner quantenchemische Modellierungs-Programme. Sie werden nun mit den hochaufgelösten neuen Datensätzen überprüft und gegebenenfalls verbessert.
GFP: Ein Alleskönner in der Biologie
In der Biologie ist GFP von großer Bedeutung – nicht umsonst bekamen die Entdecker im Jahr 2008 den Nobelpreis der Chemie verliehen. Heute ist die Verwendung von GFP als Marker für andere Proteine eine Standardmethode in der Zellbiologie. Aktuell versuchen Forschende in mehreren Studien mithilfe des GFP herauszufinden, welche Zellen das Virus SARS-Cov-2 befällt, um so die Krankheit Covid-19 besser zu verstehen.
Originalpublikation:
Shibazaki, C., Shimizu, R., Kagotani, Y., Ostermann, A., Schrader, T. E., & Adachi, M. (2019). Direct observation of the protonation states in the mutant green fluorescent protein. The Journal of Physical Chemistry Letters, 11(2), 492-496. DOI: 10.1021/acs.jpclett.9b03252
Weitere News
-
02.07.2020
Die Entgiftungsreaktion von Vitamin C
-
30.11.2016
Wasserstoffatom gesucht und gefunden
MLZ ist eine Kooperation aus:
 > Technische Universität München
> Technische Universität München > Helmholtz-Zentrum Hereon
> Helmholtz-Zentrum Hereon
 > Forschungszentrum Jülich
> Forschungszentrum Jülich
MLZ ist Mitglied in:
 > LENS
> LENS > ERF-AISBL
> ERF-AISBL
MLZ in den sozialen Medien:





