MLZ ist eine Kooperation aus:
 > Technische Universität München
> Technische Universität München > Helmholtz-Zentrum Hereon
> Helmholtz-Zentrum Hereon
 > Forschungszentrum Jülich
> Forschungszentrum Jülich
MLZ ist Mitglied in:
 > LENS
> LENS > ERF-AISBL
> ERF-AISBL
MLZ in den sozialen Medien:

MLZ
Lichtenbergstr.1
85748 Garching
BIODIFF
Diffraktometer für die Untersuchung biologischer Makromoleküle
Dieses Instrument ist auf kalte Neutronen fokussiert. Alle hier angegebenen Parameter beziehen sich auf die aktuelle Betriebsphase des FRM II. Bitte kontaktieren Sie für alle Detailfragen (Experimentdauer etc.) vorab das Instrumentteam!
Das BIODIFF ist ein monochromatisches Einkristall-Diffraktometer, das auf die Strukturbestimmung von großen biologischen Makromolekülen (oft Proteine oder DNS) hin ausgelegt ist. Bei diesen Makromolekülen spielen Wasserstoffatome eine wichtige Rolle. Sie sind z. B. verantwortlich für die Bindung des Substrats und nehmen an Protontransferreaktionen im katalytischen Zyklus vieler Enzyme teil. Daher ist es oft für ein Verständnis des zugrunde liegenden katalytischen Reaktionsmechanismus essentiell, den Protonierungszustand einzelner Aminosäure-Seitenketten im aktiven Zentrum der Enzyme zu kennen. Jedoch kann die Röntgenstrukturanalyse, welche im Allgemeinen zur Bestimmung der Proteinstruktur herangezogen wird, besonders diese oft flexiblen Wasserstoffatome im Allgemeinen nicht gut nachweisen. Die Strukturanalyse mit Neutronen hingegen erlaubt die Bestimmung der Positionen dieser Wasserstoffatome schon bei einer mittleren Auflösung von dmin < 2.5 Å.
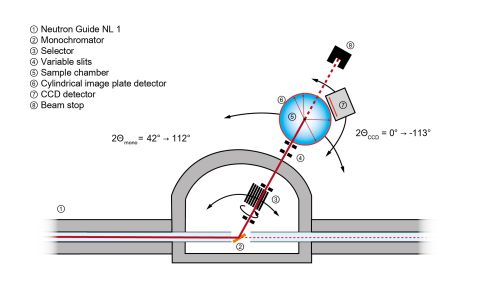
Das Instrument BIODIFF befindet sich in der Neutronenleiterhalle West am Neutronenleiter NL-1. Mittels eines Graphit-Monochromators PG(002) kann die gewünschte Wellenlänge der Neutronen kontinuierlich zwischen 2.7 Å und 4 Å aus dem weißen Strahl des Neutronenleiter als Primärstrahl ausgekoppelt werden. Die noch vorhandenen höheren Ordnungen werden durch einen Geschwindigkeitsselektor aus dem Primärstrahl entfernt. Mittels des PG(004) Reflexes kann aber auch eine Wellenlänge von 2 Å realisiert werden. Die Gitterkonstanten des Probenkristalls dürfen bei dieser Wellenlänge eine Länge von etwa 80 Å nicht überschreiten um überlappende Bragg-Reflexe zu vermeiden.
Der Hauptdetektor des Instruments ist ein Neutronen-Bildplatten-Detektor mit zylindrischer Geometrie, der etwa den halben maximalen Raumwinkel abdeckt. Als zweiter Detektor steht eine mit einem LiF/ZnS Neutronenkonverter/ Szintillator versehene CCD-Kamera zur Verfügung.
Der Hauptvorteil des Instruments BIODIFF ist, dass die verwendete Wellenlänge an die Größe der Einheitszelle und die gewünschte Auflösung angepasst werden kann. Der monochromatische Strahl hat dabei den Vorteil, dass der Untergrund geringgehalten wird, was besonders für die Messung hochaufgelöster Datensätze von Vorteil ist.
Unter den typischen Anwendungen des Instruments BIODIFF findet sich die Strukturanalyse von Proteinen mit besonderem Augenmerk auf die Positionen der Wasserstoffatome. Typische wissenschaftliche Fragestellungen sind:
- Enzymatische Mechanismen, bei denen der Protonierungszustand von bestimmten Aminosäure-Seitenketten im aktiven Zentrum eine wichtige Rolle spielt.
- Ligandenbindung, bei denen Wasserstoffbrückenbindungen involviert sind.
- Untersuchungen der Wasserstruktur an der Oberfläche oder im aktiven Zentrum der Proteinmoleküle.
- H/D-Austauschungsgrad, um die Flexibilität oder Zugänglichkeit der Proteinstruktur an verschiedenen Stellen zu beurteilen.
Neben der Standard-Probenumgebung stehen als spezielle Probenumgebungen für die Proteinkristallographie zur Verfügung:
- Ein Oxford Cryosystems Cryostream 700 plus, der einen Stickstoffgasstrom mit Temperaturen zwischen 90 und 500 K um die Probe erzeugt
- CC-Kryostat 3.5 – 325 K
- Neutronenleiter: NL-1 mit einer Nickel-Titan-Beschichtung m = 2
- Monochromatorkristall:
- Pyrolytischer Graphit (PG), Mosaizität: 0.4 – 0.5°
- Filter für höhere Ordnungen der Wellenlänge:
- Geschwindigkeits-Selektor der Firma Astrium
- Transmission 87 % für Neutronen der Wellenlänge 2.7 Å
- Verfügbarer Wellenlängenbereich:
- 2.0 Å mit PG(004): 3.0 · 106 n cm-2 s-1. Limit: Einheitszellen mit Achsen kleiner 80 Å.
- 2.7 Å mit PG(002): 4.0 · 106 n cm-2 s-1. Limit: Einheitszellen mit Achsen kleiner 110 Å.
- 3.4 Å mit PG(002): 1.8 · 106 n cm-2 s-1
- 4.0 Å mit PG(002): 1.0 · 106 n cm-2 s-1
- Durch eine Kollimation mit veränderbaren Aperturen kann der Primärstrahl auf einen Durchmesser von 1 – 4 mm eingestellt werden.
- Wellenlängen-Auflösung am Probenort: ∆λ/λ = 2.9 % bei 2.7 Å mit PG(002)
- Wellenlängen-Auflösung am Probenort: ∆λ/λ = 1.5 % bei 2.0 Å mit PG(004)
- Strahldivergenz ohne Aperturen:
- 0.8° FWHM horizontal
- 0.7° FWHM vertikal
- BaFBr: Eu2+ vermischt mit Gd2O3 als Neutronenkonverter
- Abmessungen der zylindrischen Detektortrommel:
- Radius: 200 mm
- Abgedeckter Winkelbereich:
- ± 152° horizontal
- ± 48° vertikal
- Einstellbare Pixelgröße (quadratisch): 125, 250, 500 μm
- Auslesezeit inklusive Löschen: 5 min (bei 500 μm Pixelgröße)
- ZnS vermischt mit 6LiF als Neutronenkonverter
- Abmessungen:
- Aktive Fläche des Szintillators: 200 × 200 mm²
- Abstand zur Probe: 100 mm
- Erreichbarer 2Θ-Winkel um die Probenposition: 0° – 113°
- CCD Halbleiter mit 2048 × 2048 Pixeln
- Pixelgröße (CCD-Chip): 13.5 × 13.5 μm2
- Detektor-Auflösung ≈ 300 × 300 μm2 (begrenzt durch die Dicke des Szintillatormaterials)
- Minimale Auslesezeit: ≈ 1 sec (bei voller Pixelzahl); < 1 sec (wenn mehrere Pixel auf dem Chip aufintegriert werden)
Instrumentverantwortliche
Dr. Andreas Ostermann
Telefon: +49 (0)89 289-14702
E-Mail: Andreas.Ostermann@frm2.tum.de
Dr. Tobias Schrader
Telefon: +49 (0)89 158860-743
E-Mail: t.schrader@fz-juelich.de
BIODIFF
Telefon: +49 (0)89 289-14565
Betreiber


Publikationen
Finden Sie alle aktuellen Publikationen zu BIODIFF in unserer Publikationsdatenbank iMPULSE:
Zitierung Instrument
Heinz Maier-Leibnitz Zentrum. (2015). BIODIFF: Diffractometer for large unit cells. Journal of large-scale research facilities, 1, A2. http://dx.doi.org/10.17815/jlsrf-1-19
Zitat bitte stets einschließlich DOI.
Instrumentsteuerung
Galerie









MLZ ist eine Kooperation aus:
 > Technische Universität München
> Technische Universität München > Helmholtz-Zentrum Hereon
> Helmholtz-Zentrum Hereon
 > Forschungszentrum Jülich
> Forschungszentrum Jülich
MLZ ist Mitglied in:
 > LENS
> LENS > ERF-AISBL
> ERF-AISBL
MLZ in den sozialen Medien: