MLZ ist eine Kooperation aus:
 > Technische Universität München
> Technische Universität München > Helmholtz-Zentrum Hereon
> Helmholtz-Zentrum Hereon
 > Forschungszentrum Jülich
> Forschungszentrum Jülich
MLZ ist Mitglied in:
 > LENS
> LENS > ERF-AISBL
> ERF-AISBL
MLZ in den sozialen Medien:

MLZ
Lichtenbergstr.1
85748 Garching
13.06.2017
Neutronen machen Wasserstoffatome sichtbar
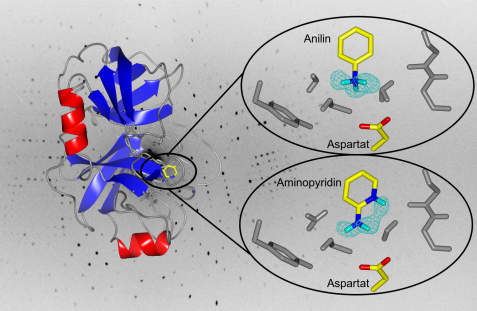
Links: das gesamte Enzym Trypsin in der Bänder-Darstellung. Rechts: Zoom in die Bindungstasche des Enzyms. Die türkise Netzstruktur stellt die gemessene Dichte für die fraglichen Wasserstoffatome des Anilin und Aminopyridin dar. Die Kopfgruppe des Anilin trägt eindeutig 3 Wasserstoffatome, ist also positiv geladen. Beim Aminopyridin weist die Kopfgruppe nur 2 Wasserstoffatome auf. Allerdings ist hier das Ring-Stickstoffatom protoniert. © A. Ostermann / FRM II, TUM
Wieder einmal hat das Instrument BioDiff am MLZ mit höchster Genauigkeit Wasserstoffatome von verschiedenen Hemmstoffen bei der Bindung an das Enzym Trypsin nachgewiesen. Die Ergebnisse, die für die Vorhersage von Bindungseigenschaften von neuen Wirkstoffen in der Arzneimittelforschung wichtig sind, wurden jetzt in der Zeitschrift Angewandte Chemie veröffentlicht.
Biomoleküle binden oft über sogenannte Wasserstoffbrücken an ihre Bindungspartner. Meist sind die dafür entscheidenden Wasserstoffatome in ihrer Position allerdings nur schwer zu bestimmen, weil Röntgenstrahlen die Wasserstoffatome nur in seltenen Fällen sichtbar machen können. Neutronen haben jedoch den Vorteil, dass sie an Atomkernen gestreut werden, hier gelten andere Gesetzmäßigkeiten bezüglich der Streustärke. Die genaue Kenntnis über die Position der Wasserstoffatome an relevanten Molekülgruppen ist jedoch entscheidend, wenn man neue Wirkstoffe in der Pharmaforschung entwickeln will. Denn die Wirkstoffe sind meist schwache Säuren oder Basen und sie wechseln leicht zwischen protoniertem Zustand (mit Wasserstoffatom) und de-protoniertem Zustand (ohne Wasserstoffatom).
Die Wissenschaftler von der Philipps-Universität Marburg und dem Istituto Italiano di Tecnologia aus Genua haben nun die Bindung der beiden Hemmstoffe (Inhibitoren) Anilin und 2-Aminopyridin an das Enzym Trypsin genauer untersucht. Trypsin diente hier als Modell-Enzym für die Klasse der so genannten Trypsin-ähnlichen Serinproteasen, die vor allem beim Verdauungsvorgang, aber auch bei der Blutgerinnung sowie an Krebs-Metastasen oder der Immunantwort beteiligt sind und bei diesen Vorgängen Proteine an spezifischen Stellen spalten. Dabei erkennt und spaltet beispielsweise Trypsin eine Aminosäurekette (das Substrat) immer nach stark basischen Aminosäuren wie Arginin oder Lysin. Für diese Spezifizität ist eine negativ geladene Aminosäure in der Bindungstasche des Trypsins verantwortlich. Substrat-ähnliche Inhibitoren mit stark basischen Kopfgruppen sind bei der Bindung an der Kopfgruppe protoniert und damit positiv geladen. Ungeklärt war jedoch, ob Bindungspartner mit einer nur schwach basischen Kopfgruppe ebenfalls im protonierten Zustand binden. Um dies zu untersuchen, wurden die beiden Inhibitoren Anilin und 2-Aminopryridin, deren chemische Eigenschaften gut charakterisiert sind, bei der Bindung an Trypsin untersucht. Anilin trägt eine schwach basische Kopfgruppe, 2-Aminopyridin ist sehr ähnlich aufgebaut, weist aber nebeneinander zwei prinzipiell protonierbare Gruppen auf.
Bei den Messungen am Neutronendiffraktometer BioDiff der Technischen Universität München und des Forschungszentrums Jülich erhielten die Marburger Forscher von TUM-Wissenschaftler Dr. Andreas Ostermann Datensätze mit der höchsten Auflösung, die bis dato für Proteine mit mehr als 100 Aminosäuren erzielt wurde. So konnten die Forscher die Protonierungszustände der Inhibitoren sehr genau bestimmen: Die Kopfgruppe des Anilin ist an der Aminogruppe außerhalb des aromatischen Rings protoniert und damit positiv geladen. Die Wissenschaftler hatten auf Grund der schwachen Basizität dies so nicht vorhergesagt. Die Kopfgruppe des 2-Aminopryridin ist stärker basisch als die des Anilins, jedoch wird dort überraschender Weise nicht die Aminogruppe am Ring bei der Bindung an Trypsin protoniert, sondern das Proton lagert sich trotz eines größeren Abstands zu der negativ geladenen Aminosäure an dem Ringstickstoff an. Für diesen Unterschied sind molekülinterne elektronische Effekte verantwortlich, die die Stabilität der Protonierungszustände bestimmen und nicht der kürzere Abstand zu der entgegengesetzten Ladung. Eine korrekte Vorhersage, wo Wirkstoffmoleküle protoniert werden und wie ihr Ladungszustand genau aussieht, ist entscheidend für die planvolle Entwicklung neuer Wirkstoffe.
Originalveröffentlichungen:
Charges shift protonation: neutron diffraction reveals that aniline and 2-aminopyridine are protonated upon binding to trypsin,
J. Schiebel, R. Gaspari, A. Sandner, K.Ngo, H-D. Gerber, A. Cavalli, A. Ostermann, A. Heine und G. Klebe
Angewandte Chemie International Edition 56, 1-5 (2017),
DOI: 10.1002/anie.20701038
und
Ladungen verschieben Protonierungen: Neutronenbeugung zeigt, dass Anilin und 2-Aminopyridin protoniert an Trypsin binden
J. Schiebel, R. Gaspari, A. Sandner, K.Ngo, H-D. Gerber, A. Cavalli, A. Ostermann, A. Heine und G. Klebe
Angewandte Chemie, 129 (17), 4965–4969 (2017)
DOI: 10.1002/ange.201701038
Weitere News
-
30.11.2016
Wasserstoffatom gesucht und gefunden
-
28.10.2015
Effektivere Nutzung von Biomasse
MLZ ist eine Kooperation aus:
 > Technische Universität München
> Technische Universität München > Helmholtz-Zentrum Hereon
> Helmholtz-Zentrum Hereon
 > Forschungszentrum Jülich
> Forschungszentrum Jülich
MLZ ist Mitglied in:
 > LENS
> LENS > ERF-AISBL
> ERF-AISBL
MLZ in den sozialen Medien:






